W tym wpisie krótko przedstawię prosty algorytm służący do grupowania punktów w zadaną liczbę klastrów.
Idea: podzielić na zadaną liczbę grup elementów, tak by najbardziej podobne do siebie były w jednej grupie. Za taką miarę podobieństwa można uznać np. bliskość położenia punktów w przestrzeni. Punkty blisko siebie uznajemy za podobne.
Odległość w przestrzeni n-wymiarowej między dwoma punktami obliczamy z:
WAŻNA UWAGA: korzystamy z algorytmu uczenia bez nadzoru, czyli algorytm sam będzie musiał wychwycić pewne zależności, w tym podejściu nie posiada on żadnych przykładów uczących. NIE mówimy tu o klasyfikacji (bo przecież nie znamy klas <- brak przykładów uczących) lecz o grupowaniu na podstawie podobieństwa, które określiliśmy wcześniej.
Jak działa ten algorytm?
1. Dla zadanego k, losuje k z pośród n punktów. (te k punkty nazywamy centroidami – punktami średnimi klastra)
2. Oblicza odległość dla każdego z n punktów do każdego z k-centroidów i przypisuje go do najbliższego klastra.
3. Dla każdego z k-klastrów zostaje wyznaczony nowy centroid jako średnia przypisanych mu elementów.
4. W pętli wykonuj od kroku 2 dopóki nie dojdziemy do momentu, że nie następują już przesunięci punktów pomiędzy klastrami
Będziemy korzystać z R struktury danych: matrix, zbioru danych iris, oraz gotowej implementacji algorytmu kmeans.
Zbiór danych iris jest jednym z przykładowych zbiorów w R. Zawiera on 150 obserwacji dla 3 rodzajów kwiatków: setosa, versicolor, virginica. Po 50 obserwacji kwiatka każdego z 3 rodzajów. Te informacje to 4 liczby dla każdej z obserwacji charakteryzujące wymiary listków oraz informacja jakiego typu był to kwiatek. Jako, że kmeans to uczenie bez nadzoru, to będziemy korzystać tylko 4 pierwszych informacji o rozmiarach.
iris
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
................................................................
summary(iris) Sepal.Length Sepal.Width Petal.Length Petal.Width Species Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100 setosa :50 1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300 versicolor:50 Median :5.800 Median :3.000 Median :4.350 Median :1.300 virginica :50 Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199 3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800 Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
Do naszych działań poręczniej będzie korzystać z macierzy zawierającej te informacje, które nas interesują niż całym data.frame iris. Dlatego tworzymy macierz o nazwie ird wskazując, które kolumny z iris mają wejść w skład. Musimy zadać liczbę kolumn macierzy.
ird<-matrix(c(iris$Sepal.Length,iris$Sepal.Width, iris$Petal.Length,iris$Petal.Width), ncol=4)
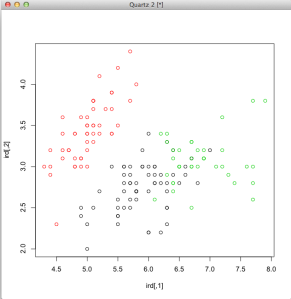
Punkty reprezentujące poszczególne obserwacje możemy zobaczyć w 2D wywołując:
plot(ird)
Wywołajmy algorytm kmeans jako argumenty podając naszą macierz i wybrane przez nas k, np.k=3
rezultaty<-kmeans(ird,3)
Stwórzmy wykres pokazujący jak pogrupowane zostały punkty
plot(ird, col=rezultaty$cluster)
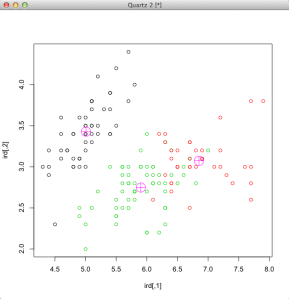
Jeśli chcemy zobaczyć wykres na którym zaznaczone zostaną nie tylko klastry, ale także finalne(z ostatniej iteracji) centroidy:
plot(ird, col=rezultaty$cluster) points(rezultaty$centers, col=6,pch=10,cex=3)
Gdybyśmy chcieli wiedzieć jakie są współrzędne każdego z 3 centroidów:
rezultaty$centers
[,1] [,2] [,3] [,4]
1 5.006000 3.428000 1.462000 0.246000
2 6.850000 3.073684 5.742105 2.071053
3 5.901613 2.748387 4.393548 1.433871
Jeśli chcemy wiedzieć ile punktów zostało przypisanych do poszczególnych grup:
table(rezultaty$cluster) 1 2 3 50 38 62